Das parodontalpathogene Mikrobiom bei Parodontitispatienten
Die ersten mikroskopischen Beobachtungen oraler Bakterien aus dem zahnadhärenten Biofilm wurden durch van Leeuwenhoek publiziert – dieses Ereignis wird allgemein als die erste orale mikrobiologische Untersuchung anerkannt [van Leeuwenhoek, 1726]. Seit dieser Zeit hat die orale Mikrobiologie zunächst Phasen des „Reduktionismus“ durchlaufen, das heißt, ein System ist verstanden, wenn man isoliert die kleinste erklärende Einheit findet, von der aus generalisiert wird. Dieser reduktionistische Ansatz geht auf den Verursacher der Tuberkulose zurück, bei dem das Vorkommen des Bakteriums Mycobacterium tuberculosis kausal eine Auslösung der Erkrankung Tuberkulose zur Folge hatte [Koch, 1882]. So ging man zunächst davon aus, dass mikrobiologisch verursachte orale Krankheiten wie Parodontitis oder Karies den Koch‘schen Postulaten folgen. In den 1970er- und 1980er-Jahren wurden zunehmend Bakterien aus an Parodontitis erkrankten Menschen isoliert und kultiviert. Bakterien wie Aggregatibacter actinomycetemcomitans und Porphyromonas gingivalis wurden auf Basis dieser Untersuchungen für die Entstehung von Parodontitis verantwortlich gemacht.
Ab den 1990er-Jahren ermöglichten molekularbiologische Nachweismethoden, beispielsweise die Polymerase-Kettenreaktion (PCR) oder die DNA-DNA-Checkerboard-Hybridisierung, den kulturunabhängigen Bakteriennachweis [Socransky et al., 1998]. Da das Genom beziehungsweise charakteristische Genomabschnitte vieler Bakterien entschlüsselt waren, konnten in einer PCR-Analyse technisch einfach und schnell Genabschnitte vervielfältigt werden, anhand derer die in der Probe vorhandenen Bakterien identifiziert wurden.
Aufgrund der hohen Sensitivität und Spezifität der molekularbiologischen Nachweise wurden potenziell parodontalpathogene Bakterien auch bei gesunden Individuen gefunden. Hieraus wuchs – im Widerspruch zu den Koch’schen Postulaten – die Erkenntnis, dass auch gesunde Patienten mit diesen Bakterien besiedelt sind und nicht zwangsläufig erkranken müssen. Dieses Wissen verlangt nach der Umsetzung einer „holistischen“ Denkweise, das heißt, dass ein System verstanden ist, wenn dessen Einbettung in ein höheres Ganzes erfolgt ist. Dem Prinzip des „Holismus“ folgend konnten so neue Konzepte wie die Interaktion zwischen Spezies, mikrobieller Gemeinschaft, Biofilmen und polymikrobiellen Erkrankungen entwickelt werden. Um die Interaktion von intraoralen Bakterien zu verstehen, ist es jedoch notwendig, das gesamte orale Mikrobiom zu kennen und sich nicht nur auf einzelne, vorher bekannte sogenannte „Leitbakterien“ zu fokussieren. Diese Identifikation des oralen Mikrobioms wird durch Sequenzierungstechnologien ermöglicht.
Sequenzierungstechnologien
Bei der DNA-Sequenzierung wird die Nukleotid-Abfolge in einem bakteriellen DNA-Molekül bestimmt. Anders als bei den zuvor erwähnten Nachweismethoden ist bei der DNA-Sequenzierungstechnologie keine Vorauswahl der Genabschnitte gesuchter Bakterien notwendig. Daher spricht man auch davon, dass Bakterien „hypothesenfrei“ nachgewiesen werden. Konnten beispielweise beim PCR-Nachweis nur die Bakterien gefunden werden, nach denen auch gesucht wurde, ermöglicht die Sequenzierungstechnologie den Nachweis von allen Bakterien, die in der Probe vorhanden waren.
Dr. med. dent. Daniel Hagenfeld

4/2005–6/2010: Studium der Zahnmedizin in Münster
6/2010–12/2010: Wissenschaftlicher Mitarbeiter, Institut für physiologische Chemie und Pathobiochemie, Universitätsklinikum Münster
1/2011–2/2012: Wissenschaftlicher Mitarbeiter, Sektion Parodontologie, Abteilung für Zahnerhaltung, Mund-, Zahn- und Kieferklinik, Universitätsklinikum Heidelberg
2/2012–3/2014: Wissenschaftlicher Mitarbeiter, Department für Infektiologie, Medizinische Mikrobiologie und Hygiene, Universitätsklinikum Heidelberg
seit 4/2014: Wissenschaftlicher Mitarbeiter, Abteilung für Parodontologie und Zahnerhaltung, Zahn-, Mund- und Kieferklinik, Universitätsklinikum Münster.
Ausgangspunkt für die Identifizierung des oralen Mikrobioms sind aus der Mundhöhle gewonnene subgingvale Plaqueproben, die Bakterienmaterial enthalten. Im Labor wird zunächst aus dem gewonnenen Probenmaterial die DNA isoliert. Dies kann mit verschiedenen kommerziellen DNA-Isolierungskits erfolgen, bei denen zunächst die Zellen mit einem Lysepuffer und unter Zuhilfenahme verschiedener Enzyme aufgeschlossen werden. Abhängig vom verwendeten System wird die DNA anschließend über Säulen mit einer DNA-Bindematrix oder mithilfe von speziellen magnetischen Partikeln von den Zelltrümmern und anderen Zellbestandteilen getrennt und gereinigt (Abbildung 1). Abschließend erfolgt die Elution der DNA von der Bindematrix mit Wasser oder einer stabilisierenden Pufferlösung.
Die so gewonnene DNA-Probe enthält ein Gemisch aus den am Probenentnahmeort vorkommenden bakteriellen Genomen, die nun über eine sogenannte „Markergenanalyse“ taxonomisch eingeordnet werden können. Goldstandard unter den Identifikationsmethoden und seit Jahren breit etabliert ist die sogenannte „16S-rRNA Gen“-basierte Identifizierung von Bakterien [Weisburg et al., 1991]. Die 16S-rRNA ist ein Bestandteil der kleinen Untereinheit prokaryotischer Ribosomen. Diese Ribosomen sind für die Proteinbiosynthese der Bakterienzelle zuständig. Da die Überlebensfähigkeit von Zellen stark mit der Fähigkeit Proteine zu synthetisieren zusammenhängt, ist die für Ribosomen codierende DNA-Sequenz größtenteils sehr selten von Mutationen betroffen. Diese hoch konservierten Gene werden bei der Teilung von Bakterien unverändert weitergegeben und sind Unterscheidungsmerkmale für Bakterienspezies. Das für das Bakterium spezifische 16S-rRNA-Gen besteht aus circa 1.550 Basenpaaren (bp), die sich in hoch konservierte, aber auch variable Bereiche, die wiederum für bestimmte Bakterienspezies charakteristisch sind, unterteilen lassen. Das Sequenzieren, also das Ablesen eines oder mehrerer dieser variablen Bereiche, und der Vergleich mit einer der verfügbaren Bakteriengenomdatenbanken ermöglichen die taxonomische Einordnung des abgelesenen Sequenzbereichs. Je nach Länge der Sequenz kann die Klassifizierung in der Regel bis auf Gattungs- oder sogar bis hinunter auf Speziesebene erfolgen.
„Next Generation Sequencing“
Ungefähr seit der Jahrtausendwende wurde die bis dahin angewandte Sanger-Sequenzierung mehr und mehr durch eine neue Generation von Sequenzierungsmethoden („Next generation sequencing“, NGS) ersetzt. Vorteil der NGS-Technologie ist eine massiv parallele Erstellung von Sequenzdaten. Anders als bei der Sanger-Sequenzierung, bei der nur wenige Sequenzen (maximal 16) parallel gelesen werden können, sind NGS-Geräte in der Lage, mehrere Millionen von Sequenzdaten (Sequenzierungsreads) gleichzeitig auszulesen. Dies führt dazu, dass selbst komplexe Proben mit vielen verschiedenen Bakterienspezies schnell und umfassend charakterisiert werden können [Loman et al., 2012].
Voraussetzung für das Ablesen der gattungs- beziehungsweise speziesspezifischen Sequenz ist die Erstellung einer sogenannten Bibliothek, die dann mit einem Sequenziergerät ausgelesen werden kann. Der erste Schritt bei der Herstellung so einer 16S-rRNA-Bibliothek ist die Vervielfältigung (Amplifikation) des 16S-rRNA-Genabschnitts, der für die Identifizierung verwendet werden soll. Wie bei der Sanger-Sequenzierung wird dies über eine PCR erreicht, in der universelle Primer eingesetzt werden, die sich in einem der konservierten Bereiche des 16S-rRNA-Gens anlagern können und so die Vervielfältigung des gewünschten Abschnitts von nahezu allen Bakterienarten ermöglichen. Aufgrund des höheren Durchsatzes kann bei der NGS-Sequenzierung aber der kostenintensive Klonierungsschritt zur Vereinzelung der 16S-rRNA-Gene aus der Patientenprobe entfallen. Im nächsten Schritt der Bibliothekserstellung für die NSG-Sequenzierung werden spezifische, an die jeweilige Technologie angepasste Adapter eingefügt, die gleichzeitig mit einem Index versehen sind. Dies ist eine Art molekularer Barcode, eine kurze einzigartige Abfolge von Nukleotiden, die die Zuordnung der einzelnen Sequenzierungsreads zur jeweiligen Ursprungsprobe und damit nicht nur die parallele Analyse aller Bakterien aus einer Probe, sondern auch eine parallele Untersuchung mehrerer Patientenproben ermöglicht. Je nach Durchsatz des verwendeten Systems können so bis zu 384 Patientenproben in einem Sequenzierungslauf analysiert werden (Abbildung 2).
Für die Sequenzierung werden die Bibliotheken der einzelnen Proben in gleichen Anteilen gemischt und dieser Pool dann dem Sequenzierer zugeführt. Die aktuell am weitesten verbreitete Methode ist das sogenannte „paired-end sequencing“ (Illumina®). Hierbei werden zwar nur vergleichsweise kurze Leseweiten zwischen 150 bp und 300 bp des insgesamt 1.550 bp langen 16S-RNA-Gens erzeugt, dafür wird das amplifizierte DNA-Fragment aber von beiden Seiten (in Vorwärts- und Rückwärtsrichtung) abgelesen. Dies kann man sich im Rahmen der Qualitätskontrolle bei der Auswertung zunutze machen. Da keine Sequenzierungstechnologie fehlerfrei arbeitet, können bei der Paired-end-Methode Sequenzierungsreads, bei denen Vorwärts- und Rückwärtssequenz nicht übereinstimmen, identifiziert und damit als fehlerbehaftete Sequenzdaten von der weiteren Analyse ausgeschlossen werden.
Einige Bakterienspezies unterscheiden sich in den 150–300 bp langen Genabschnitten nicht, daher ist mit diesen Geräten eine sichere taxonomische Klassifikation nur bis auf die Gattungsebene möglich. Beispielsweise ist bei Staphylokokken der Spezies aureus und epidermidis der hypervariable Abschnitt V4 identisch. So kann in vielen Sequenzierungsstudien, die als alleiniges Ziel die V4-Region des 16S-RNA-Gens verwenden, zwar zwischen verschiedenen Gattungen unterschieden werden, nicht aber die Spezies innerhalb einer Gattung eindeutig identifiziert werden (Abbildung 3). In den vergangenen Jahren wurden zunehmend sogenannte Long-Read-Sequenziersysteme zur Marktreife gebracht, die in der Lage sind, Sequenzen mit Leselängen von mehreren tausend Basenpaaren zu generieren. Die längere Leseweite erhöht aber auch das Risiko für potenzielle Sequenzierfehler. Diese erhöhte Fehlerrate wird zum Beispiel beim Sequel II (Pacific Biosystems®) durch die Bildung einer zirkulären Konsensussequenz, die durch das mehrfache Ablesen des vervielfältigten 16S-rRNA-Gens generiert werden kann, innerhalb der Sequenzierung wieder bereinigt (Abbildung 4).

Benjamin Ehmke
Trotzdem ist es unabhängig von der verwendeten Sequenzierungstechnologie unbedingt notwendig, das Sequenzierergebnis durch bioinformatische Verfahren und eine Reihe von Kontrollen zu überprüfen. Dabei werden die gefundenen Gen-Sequenzen von Sequenzierfehlern so gut wie möglich bereinigt. Bei der Auswertung von Mikrobiomdaten ist zu beachten, dass die Effektivität der Sequenzierung technischen und biologischen Variationen unterliegt. So können Sequenzabschnitte bestimmter Bakterienarten eventuell einfacher vervielfältigt werden als andere, oder Plaqueproben können unterschiedlich stark mit Speichel verdünnt werden. Quantitative Analysen sind aus diesem Grund sehr aufwendig, weswegen Sequenzierergebnisse oft in Prozent der Gesamtreads einer Probe angegeben werden, was wiederum die statistische Auswertung erschwert. Die meisten Sequenzen kommen nur in wenigen Proben vor. Diese seltenen Sequenzen können oft schlecht von Sequenzierfehlern unterschieden werden. Häufig müssen hunderte einzigartiger Sequenzen in einer Probe ausgewertet werden, die in ihrer Gesamtheit das Mikrobiom dieser Probe darstellen. Die Auswertung und die Interpretation der Daten stellen somit eine besondere Herausforderung und eine Art „Flaschenhals“ bei der Auswertung der NGS-Analysen dar [Knight et al., 2018].
Interpretation von NGS-Daten
Bei den klassischen mikrobiologischen und molekularbiologischen Nachweisverfahren wurde meist eine qualitative und im besten Fall eine quantitative Information zum Nachweis einzelner Bakterien in der Patientenprobe ausgegeben. So scheinbar greifbar und sinnvoll diese qualitativen und quantitativen Parameter sein mögen, geben sie jedoch keine Information, welche Rolle oder welchen Anteil an der Bakterienflora die jeweiligen Bakterien in der untersuchten Mundhöhle haben. So sind in den ökologischen Wissenschaften beispielsweise Feststellungen, ob oder wie viele Wölfe in einem Wald leben, nur wenig aussagekräftig. Die Frage, ob diese für den Menschen Schaden oder Nutzen bringen, kann nur geklärt werden, wenn untersucht wird, in welcher Gemeinschaft und in welchem Verhältnis diese mit anderen Tier- und Pflanzenarten leben. Analog verhält es sich mit der mikrobiologischen Besiedlung der Mundhöhle, das heißt dem oralen Mikrobiom. So können die Ansätze der ökologischen Wissenschaften auch für die Auswertung von NGS-Analysen oraler Plaqueproben genutzt werden. Deshalb sollten nicht mehr nur einzelne Bakterien, sondern das gesamte Ökosystem Mundhöhle betrachtet und ausgewertet werden.
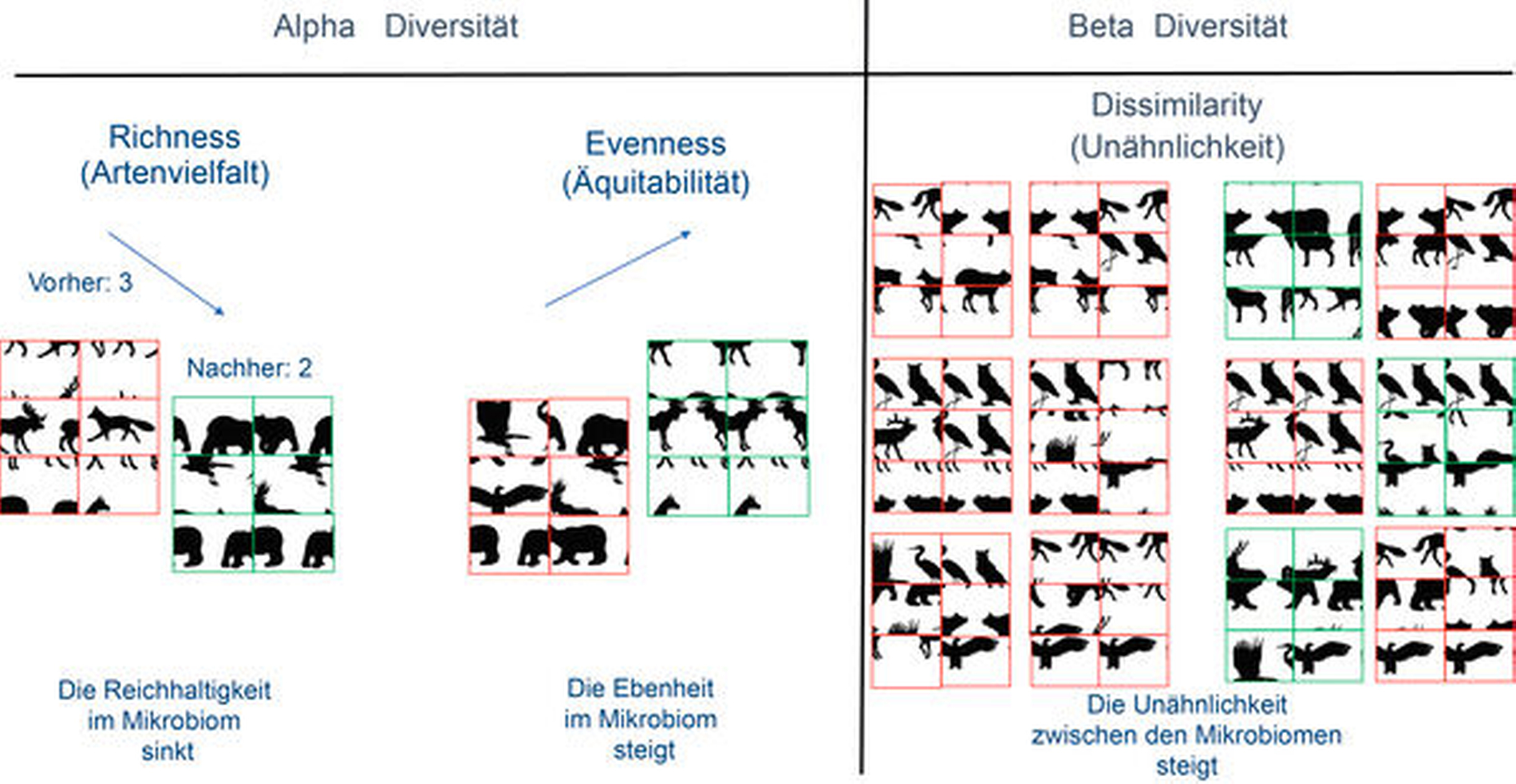
So ist es nicht verwunderlich, dass bei der Interpretation von NGS-Daten Parameter verwendet werden, die man zunächst eher in der Ökologie vermuten würde. Die sogenannte Alpha-Diversität beschreibt den Zustand eines ökologischen Systems, hier also des Mikrobioms innerhalb eines Patienten. Hierzu gehören die sogenannte „Richness“ und die „Evenness“ (Abbildung 5). Unter dem Begriff „Richness“, auch Artenvielfalt genannt, kann die Anzahl der verschiedenen Bakterien (Gattungen oder Spezies) innerhalb einer Probe verstanden werden, wohingegen die „Evenness“ die relative Häufigkeit, das heißt die Äquitabilität im Sinne der Gleichverteilung der Arten in einem Lebensraum, beschreibt. Allein die Berücksichtigung dieser beiden Parameter ermöglicht viel weitergehende Interpretationen bezüglich der parodontalen Gesundheit oder Erkrankung als die oben genannten klassischen quantitativen/qualitativen Untersuchungen von wenigen „Markerbakterien“. Die sogenannte Beta-Diversität beschreibt den Unterschied in der Artenvielfalt der in einem bestimmten Lebensraum – beispielsweise dem Ökosystem Mundhöhle – entnommenen Bakterienproben (Abbildung 5). Hier werden Distanzmatrizen der Unterschiede aller gefundenen Bakterien zueinander gebildet (zum Beispiel Bray-Curtis Dissimilarity). Dies erlaubt Abweichungen in der Mikrobiomzusammensetzung mit einer Indexzahl zu beschreiben und vereinfacht die Interpretation der Ergebnisse [Magurran, 2003].
Erkenntnisse der parodontalen Mikrobiomforschung
Erste Berichte von NGS-Studien über die Diversität des oralen Mikrobioms auf Speziesebene übertrafen die bisherigen Erkenntnisse aus den Sanger-Sequenzierungsstudien um mehrere Zehnerpotenzen [Duncan, 2005]. Schnell wurde klar, dass die NGS-Sequenzierung – wie alle anderen Methoden, die technisch vervielfältigte DNA für die Auswertung verwenden – Sequenzierfehler produziert und dass die bisherigen bioinformatischen Methoden zur Datenaufbereitung (sogenannte bioinformatische Pipelines) diese Sequenzierfehler nur unzureichend von echten Sequenzen unterscheiden konnten. Deswegen wurden Datenbanken entwickelt, die diese neu gefundenen Sequenzen überprüfen, bevor sie in die Referenzdatenbanken aufgenommen werden. Das Human Oral Microbiome Project hat sich zur Aufgabe gemacht, eine taxonomische Datenbank für orale Bakterien anzulegen [Dewhirst et al., 2010]. Diese Datenbank ist über eine Internetseite (HOMD.org) frei zugänglich und dort können bereits taxonomisch eingeordnete und noch nicht eingeordnete Gensequenzen heruntergeladen werden. Unlängst wurde diese Datenbank durch nasopharyngeale Bakterien erweitert und heißt jetzt eHOMD (expanded human oral microbiome database). Es existieren jedoch auch universelle Datenbanken mit Annotationen oraler sowie nicht oraler Bakterien [Balvočiute und Huson, 2017].
Mithilfe von Querschnittstudien wurden orale Mikrobiome von Menschen mit Parodontitis und parodontal gesunden Menschen miteinander verglichen [Abusleme et al., 2013]. Es zeigte sich, dass die bakterielle Artenvielfalt (Richness) innerhalb der Mikrobiome mit höherem Erkrankungsgrad ansteigt. Die relative Häufigkeit einiger Spezies und die Äquitabilität (Evenness) nehmen dagegen ab, so dass Mikrobiome parodontal erkrankter Menschen mehr und mehr von einzelnen Arten dominiert werden. Diese Parodontitis-Mikrobiome, die unter anderem auch die bereits bekannten parodontalpathogenen Bakterien beinhalten, sind zwischen Individuen mit Parodontitis ähnlich. Gesunde Patienten zeigen dahingegen ausgeprägte Unterschiede zueinander auf und besitzen eine sehr individuelle Mikrobiomsignatur. Interessanterweise sind diese Unterschiede bei Menschen, die im gleichen Haushalt leben und ähnliche Lebensgewohnheiten haben, geringer [Kort et al., 2014].
Diese Studien haben auch die Sicht auf die Pathogenese parodontaler Erkrankungen erweitert. So wird nicht mehr von einer spezifischen oder exogenen Infektion mit parodontalpathogenen Bakterien ausgegangen, sondern vielmehr von einer Dysbiose, das heißt von einem Ungleichgewicht in der jeweiligen oralen Flora aufgrund veränderter Umweltbedingungen [Lamont und Hajishengallis, 2015]. Die vormals parodontalpathogenen Erreger werden nun vielmehr als Pathobionten bezeichnet, deren Selektionsvorteil im oralen Mikrobiom darin besteht, beispielsweise besonders gut mit entzündlichen Veränderungen im Wirtsorganismus zurechtzukommen oder diese für die Verbreitung der eigenen Spezies zu benutzen. Vereinfacht gesagt kann beispielsweise Porphyromonas gingivalis die Blutung und erhöhte Sulkusfließrate einer leichten gingivalen Entzündung nutzen, indem die Serumproteine verstoffwechselt werden. Aus dieser verbesserten Situation heraus kann die Bakterienspezies einen größeren Raum im oralen Mikrobiom einnehmen und wird dann seinerseits eine stärkere Entzündungsreaktion im Wirt induzieren. Diese geht erneut mit verstärkter Blutung einher, was wiederum die Lebensbedingungen von Porphyromonas gingivalis zum Schaden des Patienten weiter „verbessert“.
Wie verändert die Parodontitistherapie das parodontale Mikrobiom?
Mit dem neuen Wissen aus dem Next Generation Sequencing kann auch der Einfluss der Parodontitistherapie auf ein dysbiotisches Mikrobiom anders bewertet werden. Wurden bislang nur die Veränderungen einzelner, mit der Krankheit assoziierter Bakterien erfasst, liegt nun der Fokus auf den Veränderungen des gesamten ökologischen Systems am Parodontium, respektive der Mundhöhle.
Bei Patienten mit Stadium-II/III-Parodontitis wurden innerhalb der Individuen sowohl dysbiotische als auch eubiotische (mit Gesundheit vereinbare) Mikrobiome gefunden. So waren interessanterweise intraorale Bereiche, die einem geringeren mechanischen Einfluss durch die Mundhygiene ausgesetzt waren, häufiger mit dysbiotischen Mikrobiomen besiedelt. In Plaqueproben von interdentalen und subgingivalen Entnahmestellen waren dysbiotische Mikrobiome einander ähnlicher als an supragingival bukkalen Stellen, die durch die regelmäßige tägliche Mundhygiene besser und einfacher erreicht wurden [Hagenfeld et al., 2019].
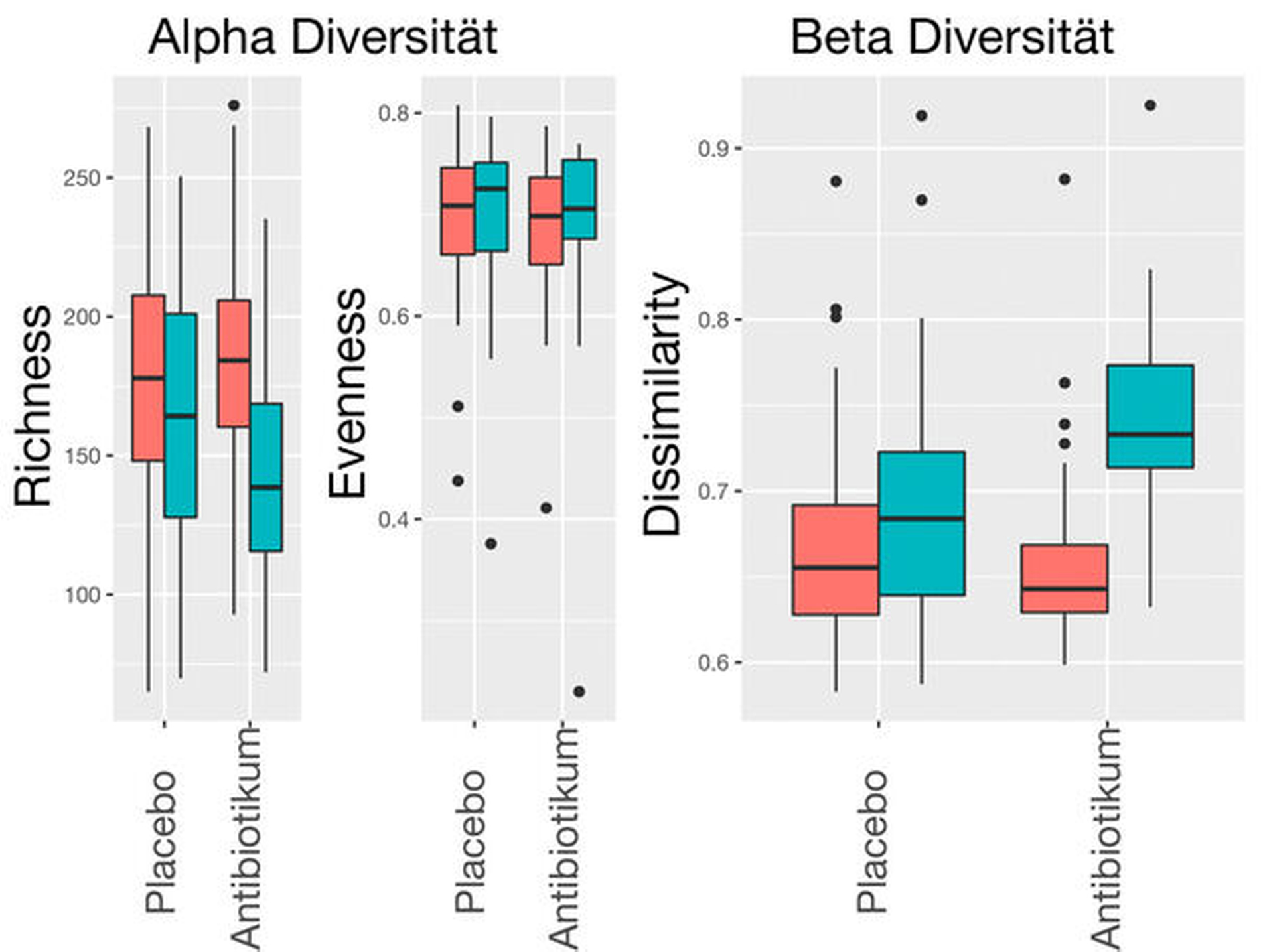
In Patienten mit Stadium-III/IV-Parodontitis nahm, verglichen mit der Ausgangsuntersuchung, die Artenvielfalt in subgingivalen Proben acht Wochen nach ausschließlich mechanischer Parodontitistherapie mit Schallscalern und Gracey-Küretten nur leicht ab (Placebogruppe, Abbildung 6). Im Gegensatz dazu konnten in dieser Studie bei Patienten, die zusätzlich zur mechanischen Therapie eine systemische Antibiotikabehandlung über sieben Tage mit 400 mg Metronidazol und 500 mg Amoxicillin erhielten, signifikante Mikrobiomveränderungen nach acht Wochen gefunden werden [Hagenfeld et al., 2018]. Die Richness im subgingivalen Mikrobiom war signifikant gegenüber dem Ausgangsbefund und der Kontrollgruppe reduziert (Antibiotikagruppe, Abbildung 6), was mit einer abnehmenden Artenvielfalt interpretiert werden kann. Weiter zeigte sich die deutliche Reduktion der Bakteriengenera, die potenziell parodontalpathogene Bakterien wie Porphyromonas ssp. und Tannerella ssp. enthalten. Gleichzeitig wurde ein Anstieg von unter anderem Actinomyceten ssp. gefunden, die eher bei parodontal gesunden Patienten zu den dominierenden Gattungen gehören. Die Evenness stieg an, was im Sinne einer gleichmäßigeren Häufigkeitsverteilung der Bakteriengenera im subgingivalen Mikrobiom interpretiert werden kann. Zusätzlich zeigte eine deutlich stärkere Veränderung der Beta-Diversität in der Antibiotikagruppe, dass sich in dieser Gruppe mehr Mikrobiome in ihrer Zusammensetzung signifikant von den Ausgangsmikrobiomen unterschieden. Sind die Mikrobiomveränderungen nach Parodontitistherapie mit adjuvanter systemischer Antibiose auch stärker ausgeprägt, lassen sich diese aber ebenfalls in der ausschließlich mit mechanischer Therapie behandelten Gruppe erkennen. Diese Mikrobiomveränderungen waren in beiden Behandlungsgruppen mit einer, in der Antibiotikagruppe zwar ausgeprägteren, aber auch in der Scalinggruppe gleichsam deutlichen und klinisch relevanten Verbesserung der klinischen Parameter verbunden.
Zudem konnte in einer weiteren Studie herausgefunden werden, dass die Zusammensetzung des Ausgangsmikrobioms vor der Therapie einen größeren Einfluss auf die Verbesserung der klinischen Parameter nach der Therapie hatte als die Tatsache, ob ein Antibiotikum oder eine Placebo-Medikation verabreicht wurde [Bizzarro et al., 2016]. Interessanterweise waren die mikrobiellen Effekte der Parodontitistherapie bei aktiven Rauchern weitgehend identisch zu den beobachteten Ergebnissen bei Nichtrauchern. Raucher zeigen aber oft Krankheitsrezidive und die mechanische Parodontitistherapie ist weniger erfolgreich als bei Nichtrauchern [Hagenfeld et al., 2020]. Raucher zeigten eine schnellere Rekolonisation mit dysbiotischer Mikroflora als Nichtraucher [Joshi et al., 2014]. Die antimikrobielle mechanische Therapie könnte – gegebenenfalls ergänzt durch eine siebentägige systemische Antibiose – somit eine Chance für das Parodontium des Patienten darstellen, eine Phase der mikrobiell ungestörten posttherapeutischen parodontalen Reparation einzugehen. Die unausweichliche Neubesiedlung nach Ende der Antibiotikatherapie würde dann bei entsprechend prädisponierten Patienten nicht wieder erneut eine dysbiotische Entwicklung nehmen, sondern zu einer Rebiose führen.
Perspektiven des NGS in der Parodontologie
Bislang wird im Bereich der Zahnmedizin das NGS nur zu wissenschaftlichen Zwecken durchgeführt. Wenngleich die Aufbereitung der Mikrobiomproben im Labor kein Problem darstellt, erschweren der schon oben erwähnte Engpass bei der bioinformatorischen Aufbereitung und die bislang fehlende Standardisierung auf diesem Gebiet einen Einsatz in der Patientenroutine. Jedoch ist fest davon auszugehen, dass das NGS in die zahnärztliche Diagnostikroutine Einzug halten wird. Im Sinne einer personalisierten Medizin könnten mit der NGS-Technologie bereits Patienten mit dysbiotischem Mikrobiom identifiziert werden, bevor diese die eigentlichen klinischen Krankheitssymptome, Taschensondierungstiefen und Attachmentverluste, zeigen. Dies würde es ermöglichen mit der Parodontitistherapie früher und weniger invasiv zu beginnen.
Eine weitere Behandlungsmöglichkeit bestünde in der Transplantation eines eubiotischen Mikrobioms in Patienten mit dysbiotischem Mikrobiom [Pozhitkov et al., 2015]. Die Therapieoption wird in der Inneren Medizin schon verschiedentlich angewendet. Beides, früherer Behandlungsbeginn und/oder Mikrobiomtransplantation, könnten dazu führen, dass das Auftreten der Parodontitis bis in ein höheres Lebensalter verzögert wird. Möglicherweise kann künftig die Bestimmung des Grades der Dysbiose für die Findung von Parametern zum Festlegen der Behandlungsfrequenz in der primären und in der sekundären Prophylaxe genutzt werden. Dies würde im Sinne einer individualisierten personalisierten Medizin Über- und Untertherapie vermeiden.
Zuletzt könnte eine Mikrobiomauswertung vor dem Einsatz adjuvanter Antibiotika in der Parodontitistherapie in Zukunft dabei helfen, diejenigen Patienten zu identifizieren, die von der Antibiose klinisch relevant profitieren. Hierdurch würde der ungerechtfertigte Einsatz von Antibiotika in der Zahnmedizin weiter reduziert werden. Viele dieser Überlegungen sind Gegenstand der aktuellen Forschung und die kommenden Jahre werden zeigen, welche Erkenntnisse für die klinische Routine relevant sein werden.
Literaturliste
Abusleme, L., Dupuy, A. K., Dutzan, N., Silva, N., Burleson, J. A., Strausbaugh, L. D., et al. (2013). The subgingival microbiome in health and periodontitis and its relationship with community biomass and inflammation. ISME J. 7, 1016–25. doi:10.1038/ismej.2012.174 .
Balvočiute, M., and Huson, D. H. (2017). SILVA, RDP, Greengenes, NCBI and OTT - how do these taxonomies compare? BMC Genomics 18. doi:10.1186/s12864-017-3501-4 .
Bizzarro, S., Laine, M. L., Buijs, M. J., Brandt, B. W., Crielaard, W., Loos, B. G., et al. (2016). Microbial profiles at baseline and not the use of antibiotics determine the clinical outcome of the treatment of chronic periodontitis. Sci. Rep. 6, 20205. doi:10.1038/srep20205 .
Chial, H. (2008). DNA sequencing technologies key to the Human Genome Project. Nat. Educ. 1, 219.
Dewhirst, F. E., Chen, T., Izard, J., Paster, B. J., Tanner, A. C. R., Yu, W. H., et al. (2010). The human oral microbiome. J. Bacteriol. 192, 5002–5017. doi:10.1128/JB.00542-10. Duncan, M. J. (2005). Oral microbiology and genomics. Periodontol. 2000 38, 63–71. doi:10.1111/j.1600-0757.2005.00111.x. Hagenfeld, D., Koch, R., Jünemann, S., Prior, K., Harks, I., Eickholz, P., et al. (2018). Do we treat our patients or rather periodontal microbes with adjunctive antibiotics in periodontal therapy? A 16S rDNA microbial community analysis. PLoS One 13. doi:10.1371/journal.pone.0195534 .
Hagenfeld, D., Matern, J., Prior, K., Harks, I., Eickholz, P., Lorenz, K., et al. (2020). Significant short-term shifts in the microbiomes of smokers with periodontitis after periodontal therapy with amoxicillin & metronidazole as revealed by 16S rDNA amplicon next generation sequencing. Front. Cell. Infect. Microbiol., under review.
Hagenfeld, D., Prior, K., Harks, I., Jockel-Schneider, Y., May, T. W., Harmsen, D., et al. (2019). No differences in microbiome changes between anti-adhesive and antibacterial ingredients in toothpastes during periodontal therapy. J. Periodontal Res. doi:10.1111/jre.12645 .
Joshi, V., Matthews, C., Aspiras, M., de Jager, M., Ward, M., and Kumar, P. (2014). Smoking decreases structural and functional resilience in the subgingival ecosystem. J. Clin. Periodontol. 41, 1037–1047. doi:10.1111/jcpe.12300 .
Knight, R., Vrbanac, A., Taylor, B. C., Aksenov, A., Callewaert, C., Debelius, J., et al. (2018). Best practices for analysing microbiomes. Nat. Rev. Microbiol. 16. doi:10.1038/s41579-018-0029-9. Koch, R. (1882). Die Ätiologie der Tuberkulose. (Nach, einem in der Physiologischen Gesellschaft zu Berlin am 24. März 1882 gehaltenen Vortrage.). Available at: https://edoc.rki.de/bitstream/handle/176904/5163/428-445.pdf?sequence=1&isAllowed=y [Accessed December 22, 2020] .
Kort, R., Caspers, M., Graaf, A. Van De, Egmond, W. Van, Keijser, B., and Roeselers, G. (2014). Shaping the oral microbiota through intimate kissing. 2, 1–8. doi:10.1186/2049-2618-2-41. Lamont, R. J., and Hajishengallis, G. (2015). Polymicrobial synergy and dysbiosis in inflammatory disease. Trends Mol. Med. 21, 172–183. doi:10.1016/j.molmed.2014.11.004 .
Loman, N. J., Constantinidou, C., Chan, J. Z. M., Halachev, M., Sergeant, M., Penn, C. W., et al. (2012). High-throughput bacterial genome sequencing: An embarrassment of choice, a world of opportunity. Nat. Rev. Microbiol. 10, 599–606. doi:10.1038/nrmicro2850. Magurran, A. E. (2003). Measuring Biological Diversity - Anne E. Magurran. 1st ed. Wiley-Blackwell.
Pozhitkov, A. E., Leroux, B. G., Randolph, T. W., Beikler, T., Flemmig, T. F., and Noble, P. A. (2015). Towards microbiome transplant as a therapy for periodontitis: An exploratory study of periodontitis microbial signature contrasted by oral health, caries and edentulism. BMC Oral Health 15. doi:10.1186/s12903-015-0109-4 .
Sanger, F., Nicklen, S., and Coulson, A. R. (1977). DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74, 5463–5467. doi:10.1073/pnas.74.12.5463 .
Socransky, S. S., Haffajee, A. D., Cugini, M. A., Smith, C., and Kent, R. L. (1998). Microbial complexes in subgingival plaque. J. Clin. Periodontol. 25, 134–44. doi:10.1111/j.1600-051X.1998.tb02419.x. van Leeuwenhoek, A. (1726). The Select Works of Anthony Van Leeuwenhoek: Containing His Microscopical ... Google Books. Available at: https://books.google.de/books?hl=de&lr=&id=qcQzAQAAMAAJ&oi=fnd&pg=PA101&dq=leeuwenhoek&ots=6-BHbYemYZ&sig=HthxS0zpYU3O9d0D3tA-d5JNzz0#v=onepage&q=leeuwenhoek&f=false [Accessed December 22, 2020] .
Weisburg, W. G., Barns, S. M., Pelletier, D. A., and Lane, D. J. (1991). 16S ribosomal DNA amplification for phylogenetic study. J. Bacteriol. 173, 697–703. doi:10.1128/jb.173.2.697-703.1991 .