Molekül aus Neandertaler-Zahnstein zum „Leben“ erweckt
„Es ist uns erstmals gelungen, Substanzen neu herzustellen, die vor hunderttausend Jahren von Bakterien produziert wurden – die Paläofurane“, berichtet der Chemiker Pierre Stallforth, Abteilungsleiter am Leibniz-Institut für Naturstoff-Forschung und Infektionsbiologie (Leibniz-HKI). Ziel des Forschungsverbunds um Stallforth ist, bereits ausgestorbene Bakterien als Quelle für neue Arzneimittel zu erschließen. Mikroben gelten als „die größten Chemiker der Natur“, und durch die Untersuchung der Genome uralter Bakterien ließen sich möglicherweise neue Verwendungsmöglichkeiten für sehr alte Moleküle entdecken, so die Hoffnung der Forscher.
Ausgestorbene Bakterien als Quelle für neue Arzneimittel
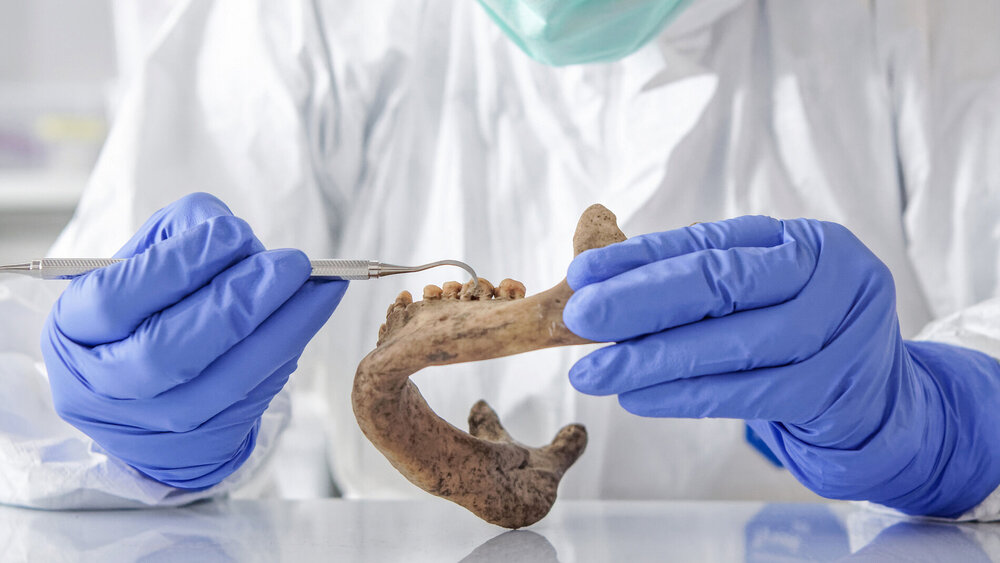
Als Grundlage dient Bakterien-DNA: Um an die DNA von steinzeitlichen Mikroorganismen zu kommen, nutzte das Team Zahnstein von Neandertalern, die vor circa 100.000 bis 40.000 Jahren lebten und von Menschen, die vor 30.000 bis 150 Jahren lebten. Mithilfe bioinformatischer Methoden rekonstruierten die Forschenden daraus die Genome zahlreicher Bakterienarten.
„Die große Herausforderung lag darin, Fehler in der abgebauten DNA zu beheben und Verunreinigungen zum Beispiel durch jüngere DNA auszuschließen“, sagt Anan Ibrahim, Postdoktorandin am Leibniz-HKI und Erstautorin der Studie. Neben vielen Bakterien, die auch heute noch die menschliche Mundflora besiedeln, fand sie ein unbekanntes Mitglied der Gattung Chlorobium. Dessen stark geschädigte DNA wies alle Merkmale eines fortgeschrittenen Alters auf und wurde im Zahnstein von sieben steinzeitlichen Menschen und Neandertalern gefunden. Alle sieben Chlorobium-Genome enthielten ein Biosynthese-Gencluster mit unbekannter Funktion.

Bei den stark abgebauten, extrem kurzen DNA-Fragmenten aus der Steinzeit eine große Herausforderung: Nach drei Jahren haben die Forscher endlich einen Durchbruch erzielt. Es gelang, DNA-Abschnitte mit einer Länge von mehr als 100.000 Basenpaaren zu rekonstruieren und eine Vielzahl alter Gene und Genome wiederherzustellen.© Werner Siemens Foundation, Felix Wey
Zahnstein der etwa 19.000 Jahre alten „Roten Dame von El Mirón“
Ein besonders gut erhaltenes Chlorobium-Genom wurde aus dem Zahnstein der etwa 19.000 Jahre alten „Roten Dame von El Mirón“, Spanien, rekonstruiert. Das 2010 in einer spanischen Höhle gefundene Skelett ist der älteste Beleg für eine Beisetzung in der Epoche des Magdalénien auf der Iberischen Halbinsel.
„Nachdem wir diese rätselhaften alten Gene entdeckt hatten, wollten wir herauszufinden, was sie bewirken“, erläutert Ibrahim. Mit biotechnologischen Methoden gelang es den Forschenden, die Gene in lebende Bakterien einzubauen, die daraus tatsächlich funktionale Enzyme bildeten.
Damit sind sie die ersten, die diesen Ansatz erfolgreich auf Zehntausende Jahre alte Bakterien-DNA anwenden. Die reaktivierten Enzyme produzieren wiederum eine neue Familie mikrobieller Naturstoffe, die die Forschenden „Paläofurane“ genannt haben. „Das ist der erste Schritt, um die verborgene chemische Vielfalt der Mikroben der Erdgeschichte zu erschließen“, sagt Martin Klapper, Postdoktorand am Leibniz-HKI und ein weiterer Erstautor der Studie.
Christina Warinner, Associate Professor an der Harvard University und Gruppenleiterin am Max-Planck-Institut für evolutionäre Anthropologie (MPI-EVA) ergänzt: „Mit dieser Studie haben wir einen wichtigen Meilenstein erreicht, um die enorme genetische und chemische Vielfalt unserer mikrobiellen Vergangenheit aufzudecken.“
Der erste Schritt, um die Mikroben der Erdgeschichte zu erschließen
Dieser Erfolg ist das Ergebnis einer einzigartigen Zusammenarbeit von Forschenden aus Archäologie, Bioinformatik, Molekularbiologie und Chemie: „Mit der Förderung durch die Werner Siemens-Stiftung wollen wir eine Brücke zwischen den Geistes- und Naturwissenschaften schlagen. Sie hat uns den Aufbau des neuen Forschungsbereichs Paläobiotechnologie ermöglicht“, sagt Stallforth. Warinner: „So konnten wir Technologien entwickeln, um Moleküle neu entstehen zu lassen, die bereits vor hunderttausend Jahren produziert wurden.“ Für die Zukunft hofft das Team, diese Herangehensweise zur Suche nach neuen Antibiotika einsetzen zu können.
Neben der Unterstützung durch die Werner Siemens-Stiftung wurde die Arbeit auch durch die Max-Planck-Gesellschaft, die Leibniz-Gemeinschaft und durch die Deutsche Forschungsgemeinschaft im Rahmen des Exzellenzclusters Balance of the Microverse und des Sonderforschungsbereichs ChemBioSys gefördert.
Originalstudie: Klapper M, Hübner A, Ibrahim A, Wasmuth I, Borry M, Haensch VG, Zhang S, Al-Jammal WK, Suma H, Fellows Yates JA, Frangenberg J, Velsko IM, Chowdhury S, Herbst R, Bratovanov EV, Dahse H-M, Horch T, Hertweck C, González Morales MR, Straus LG, Vilotijevic I, Warinner C, Stallforth P (2023). Natural products from reconstructed bacterial genomes of the Middle and Upper Paleolithic. Science, https://www.doi.org/10.1126/science.adf5300